Aktuelles
Einladung zum
Freiburg Internal Extracellular Vesicles Meeting – Get Together EV Researchers and EV-Interested
Freitag, 25. Oktober 2024.

Eigene Darstellung
Aktionstage „Nicht jeder Handgriff benötigt Handschuhe“
In den vergangenen Jahren hat der Gebrauch unsteriler Handschuhe in Situationen, in denen eine Verwendung gar nicht notwendig ist, enorm zugenommen.
Unter dem Motto „Nicht jeder Handgriff benötigt Handschuhe“ informierten wir vom 04. bis 10. Juli klinikumsweit zu diesem Thema und kamen an unseren festen und mobilen Infoständen insbesondere mit vielen in der direkten Patientenversorgung tätigen Mitarbeiter*innen ins Gespräch.
Ziel war und ist, aufzuklären und zu sensibilisieren, in welchen Fällen es tatsächlich notwendig ist, sich mit Handschuhen zu schützen und in welchen Situationen Handschuhe nicht erforderlich bzw. aus infektionspräventiver oder arbeitsmedizinischer Sicht (Stichwort Hautschutz) sogar bedenklich sind.
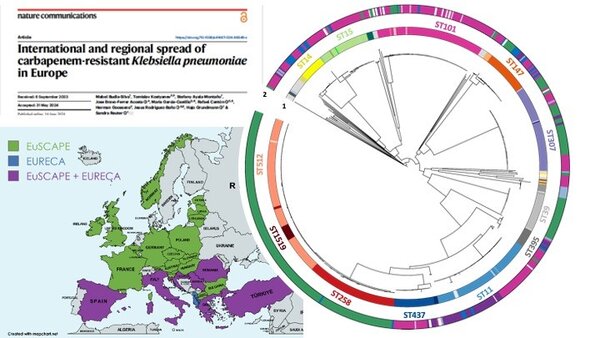
Nature Communications: Internationale und regionale Verbreitung von Carbapenem-resistenten Klebsiella pneumoniae in Europa
Antibiotika-Resistenzen stellen Krankenhäuser vor große Herausforderungen, da sie die Therapie-Optionen einschränken. Dies gilt besonders für Carbapeneme, eine Gruppe von Breitband-Antibiotika, die oft als letzte Möglichkeit eingesetzt werden, um schwere bakterielle Infektionen zu behandeln, die beispielsweise durch Klebsiella pneumoniae hervorgerufen werden.
Ein internationales Konsortium – COMBACTE-CARE EURECA – unter der Leitung von Dr. Sandra Reuter hat nun in einer Studie die Carbapenem-resistenten Varianten von Klebsiella pneumoniae untersucht, die in Südeuropa zirkulieren. Durch den Vergleich mit früheren Studien ist es so möglich, zeitliche Veränderungen aufzuzeigen. So ist zum Beispiel ersichtlich, dass es in der Türkei jetzt eine besondere Variante gibt, die gleich mehrere Resistenzmechanismen trägt. Weitere regionale Unterschiede finden sich in einer Variante in Serbien, die jetzt resistenter ist als in den vergangenen Jahren.
Die Studie untersuchte ebenfalls die vorkommenden Resistenzmechanismen in den verschiedenen Ländern und Varianten. Von besonderer Besorgnis ist eine Variante, die sich besonders leicht anpassen kann, und verschiedene Resistenzmechanismen je nach Land aufweist.
Budia Silva, M., Kostyanev, T., Ayala Montaño, S., Bravo-Ferrer Acosta, J., Garcia-Castillo, M., Cantón, R., Goossens, H., Rodriguez-Baño, J., Grundmann, H., and Reuter, S. (2024). International and regional spread of carbapenem-resistant Klebsiella pneumonia in Europe. Nat Comm 15(1): 5092. https://doi.org/10.1038/s41467-024-49349-z

Eigene Darstellung
Das Institut bekommt ein neues Gesicht
Analyse- und Vorhersagetool für COVID-19
Während der COVID-19-Pandemie entwickelte unsere Arbeitsgruppe Quantitative und Prädiktive Infektionsepidemiologie Modelle zur Vorhersage des Bettenbedarfs, die für die Pandemieplanung des Krankenhauses unerlässlich waren. Diese Modelle wurden so verallgemeinert, dass sie auf jedes deutsche Krankenhaus passen, je nachdem, wo die meisten der aufgenommenen COVID-19-Patienten leben. Wir haben diese Modelle dann als kostenloses Online-Tool öffentlich zugänglich gemacht. Das Tool wird laufend mit den neuesten verfügbaren Daten zur regionalen Inzidenz und Bettenbelegung aktualisiert. Dieses Projekt war das Ergebnis einer engen Zusammenarbeit mit den Universitätskliniken in Tübingen, Ulm, Heidelberg und Mannheim.
Analyse- und Vorhersagetool für COVID-19