Warum lohnt sich die Zusammenarbeit mit der FREEZE-Biobank?
Professionelles Biobanking erleichtert Ihnen das Leben!
Warum das so ist, erfahren Sie in diesem Informationsfilm der Biobank-Community in D-A-CH (German Biobank Node, BBMRI.at und Swiss Biobanking Platform):
Quelle: GBN - BBMRI.at - Swiss Biobanking Platform
Proben-Anfrage
Wie kann ich Bioproben aus der FREEZE-Biobank anfordern?
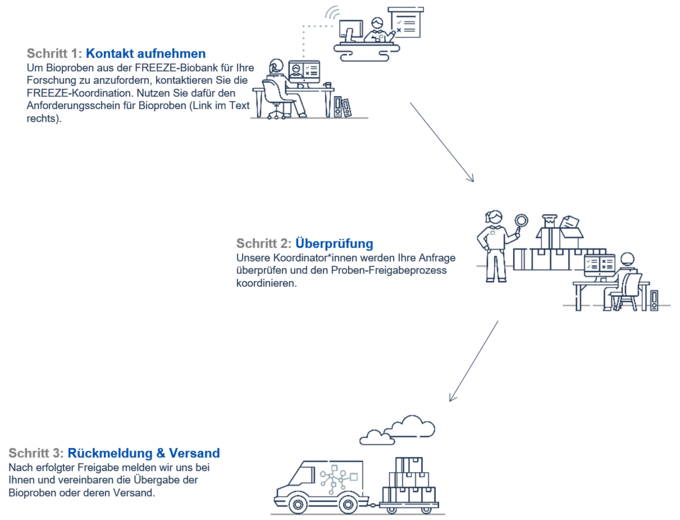
- Kontakt aufnehmen
Um Bioproben aus der FREEZE-Biobank für Ihre Forschung anzufordern, nutzen Sie bitte den Anforderungsschein für Bioproben aus der FREEZE-Biobank (English Version here). Den vollständig ausgefüllten und unterschriebenen Anforderungsschein senden Sie bitte per E-Mail an das FREEZE-Koordinationsteam. Besonderes Augenmerk wird auf das Vorliegen eines positiven Votums der für das Forschungsprojekt zuständigen Ethik-Kommission gelegt.
Alle Regelungen zur Probenanforderung bei der FREEZE-Biobank sind in der FREEZE-Nutzungsordnung festgehalten. Diese lassen wir Ihnen auf Anfrage sehr gerne zukommen (Mailto: FREEZE-Koordinationsteam). - Überprüfung
Unser Koordinationsteam wird Ihre Anfrage überprüfen. Dazu werden die folgenden Punkte betrachtet:- Vorliegen der Proben in der gewünschten Qualität und Quantität
- Überprüfung der Einwilligung
- Koordination der Probenfreigabe durch das zuständige Wissenschaftliche Vergabekomitee
- Rückmeldung & Versand
Wir melden uns bei Ihnen zum nächstmöglichen Zeitpunkt für Rückfragen oder direkt zur Vereinbarung eines Termins zur Übergabe oder zum Versand der Bioproben.
Projekt-Anfrage
Wie kann ich mit der FREEZE-Biobank eine Bioprobensammlung aufbauen?
- Kontaktaufnahme
Um in Zusammenarbeit mit der FREEZE-Biobank eine Kohorte oder eine Probensammlung für eine Studie aufzubauen, nutzen Sie bitte den Antrag Kohortenaufbau-Studien FREEZE-Biobank. Den vollständig ausgefüllten und unterschriebenen Antrag senden Sie bitte per E-Mail an das FREEZE-Koordinationsteam. - Überprüfung und Projektbesprechung
Unser Koordinationsteam wird Ihre Anfrage überprüfen und zum nächstmöglichen Zeitpunkt Kontakt mit Ihnen aufnehmen, um in einer oder mehreren Projektbesprechungen weitere Details zu klären. Hierbei werden folgende Themen beleuchtet:- Sammelstrategie
- Probentransport
- Verarbeitungsprotokolle
- Besonderheiten bei der Dokumentation
- Einwilligungsmanagement
- ggf. Ethikantrag (beratend)
- Finanzierung
- Bereitstellung der Projektdokumente
Auf Basis der Projektbesprechungen wird das FREEZE-Koordinationsteam die Projektdokumente zusammenstellen und mit Ihnen final abstimmen:- Projektplan
- Kostenkalkulation
- Nutzungsvertrag
- Projektstart
Sie können mit der Einsendung der Bioproben beginnen. - Rückmeldung
Nach einer individuell festzulegenden Pilotphase erfolgt eine Rückmeldung durch das FREEZE-Koordinationsteam, ggf. gefolgt von Anpassungen
Leistungsverzeichnis
- Bereitstellung von Bioproben und Daten, welche von der anfordernden Person zuvor in die FREEZE-Biobank eingebracht wurden
- Bereitstellung von Bioproben und Daten, ohne dass diese von der anfordernden Person zuvor selbst in die FREEZE-Biobank eingebracht wurden
- Prozessierung von Bioproben gemäß Tabelle 1 in den Räumlichkeiten der FREEZE-Biobank
- Lagerung von Bioproben in den Räumlichkeiten der FREEZE-Biobank
- Schulung zur Bioprobenverarbeitung nach den Vorgaben der FREEZE-Biobank
- Anbindung von historischen Probensammlungen an die FREEZE-Biobank (detaillierte Beschreibung in § 6 und § 7 der Nutzungsordnung)
-
Tabelle 1: Angebotene Probenarten:
Stammprobe Probenart Volumen / Konzentration Langzeit-Lagerung Vollblut (ohne Antikoagulanz) Serum 0.5 ml < -175°C Vollblut (EDTA, Citrat, Heparin) Plasma 0.5 ml < -175°C Vollblut 0,5 - 1ml -80°C Buffy Coat 0.5 ml -80°C PBMCs (Peripheral Blood Mononuclear Cells) 1 ml, 9-17 x 106/ml < -175°C Knochenmark (EDTA, Heparin) BMMCs (Bone Marrow Mononuclear Cells) 1 ml, 9-17 x 106/ml < -175°C Knochenmark 1 ml Urin Urin-Überstand 1.5 ml -80°C Urin-Sediment 0.5 ml -80°C Stuhl (stabilisiert) Stuhl (stabilisiert) 1.5 ml -80°C RNA-stabilisiertes Vollblut (PAXgene™) RNA-stabilisiertes Vollblut (PAXgene™) Originalröhrchen -80°C Galle Voll-Galle 1.5 ml -80°C Galle-Überstand 1.5 ml -80°C Galle-Sediment 0.5 ml -80°C Milz SMC (Spleen Mononuclear Cells) 1 ml, 17-25 x 106/ml < -175°C Speichel Speichel Originalröhrchen -80°C
- Betrieb und Administration des zentralen Biobank-Informations-Management-Systems (BIMS) CentraXX
- Bereitstellung von CentraXX für die Hubs
- Benutzer*innenschulung CentraXX
- Einrichtung von Organisationseinheiten
- Anbindung von CentraXX an das Datenintegrationszentrum (Einrichtungsphase 07/22)
Beratung zum Einwilligungsmanagement
- Management und Dokumentation der FREEZE-Patient*innen- und Proband*innen-Einwilligungserklärungen
- Bereitstellung von Informationen über den Einwilligungsstatus von Patient*innen und Proband*innen
- Umsetzung des Widerrufsrechts und anderer Einwilligungs-bezogener Patient*innen- und Proband*innen-Rechte
Beratung zur Erstellung von QM-Dokumenten (Verfahrensanweisungen, Arbeitsanweisungen, Formulare) zu Biobanking-Prozessen nach den Vorgaben der FREEZE-Biobank
Einwilligungs-Szenarien (mit der Ethik-Kommission abgestimmt):
Szenario Nr. Kurzbegriff Zusatz Beschreibung Aufklärung/Einwilligung 1
Allgemeines Biobanking
(Ziel: prospektiver Kohortenaufbau)
- ohne
Bioproben werden zum Zweck des Kohortenaufbaus für noch nicht bekannte zukünftige Forschungsprojekte langfristig in der Biobank eingelagert.
Ggf. wiederholte Bioprobeneinlagerung im Rahmen von Kontrollterminen
FREEZE-BC
Re-Konsentierung mindestens alle 5 Jahre
- plus Rest- oder Extra-Frischproben
(1) Bioproben werden zum Zweck des Kohortenaufbaus für noch nicht bekannte zukünftige Forschungsprojekte in der Biobank eingelagert
(2) und parallel dazu sollen für ein konkretes Forschungsprojekt frische Rest- oder Extra-Proben sofort verwendet werden (Sensitive Technologien wie z.B. Single Cell Technologien, die den Einsatz von Frischproben erfordern).
Biobanking muss im Vordergrund des Interesses stehen und der Einsatz von Frischgewebe für die unmittelbare Forschung darf nicht den Hauptfokus der angestrebten Sammlung darstellen.
(1) FREEZE-BC
(2) Beiblatt Frischproben mit Unterschrift Patient*in inkl. Infoblatt mit Projektbeschreibung(en) des Projekts/der Projekte, für das/die die Frischproben eingesetzt werden können
(Templates für Beiblatt und Infoblatt werden von FREEZE auf Anfrage zur Verfügunge gestellt.)
2
Studien-spezifisches Biobanking
- nur für Studie
Prozessierung und Einlagerung von Bioproben für eine Studie als reines Auftragsbiobanking.
Es werden KEINE Bioproben für eine offene spätere Verwendung eingelagert, sondern lediglich die Proben zu einem späteren Zeitpunkt für die studien-spezifischen Analysen wieder herausgegeben. Es werden alle Proben für die Studie aufgebraucht oder übrige Proben vernichtet.
Studien-spezifischer ausführlicher IC,
incl. Beschreibung der Studien-spezifischen Bioprobenverarbeitung und -lagerung in der Biobank (FREEZE-Biobank als reine Service-Einheit)
- für Studien zu Therapie oder Diagnostik, von denen der/die Teilnehmer*in einen persönlichen oder auch Gruppen-Nutzen erwarten kann und zusätzlich breite Verwendung der Proben
(1) Prozessierung und Einlagerung von Bioproben für eine Studie.
(2) Zusätzlich optional Einlagerung von Bioproben für noch nicht bekannte zukünftige Forschungsprojekte in der Biobank (Allgemeines Biobanking).
Aufklärung:
(1) Studien-spezifischer ausführlicher IC
(2) optional FREEZE-BC
Einwilligung:
FREEZE-BC nicht Voraussetzung für Studienteilnahme von Patient*innen – die Bereitschaft zur Teilnahme am Biobanking darf nicht darüber entscheiden, ob Patient*innen eine Studienteilnahme und insbesondere Therapiemöglickeit offen steht oder nicht.
- für Studien zu Therapie oder Diagnostik, von denen der/die Teilnehmer*in einen persönlichen oder auch Gruppen-Nutzen erwarten kann und evtl. später breite Verwendung der Proben
(1) Prozessierung und Einlagerung von Bioproben für eine Studie.
(2) Im Falle dass NICHT alle Proben für die Studie aufgebraucht werden, sollen die übrigen Studienproben nachträglich optional dem allgemeinen Biobanking zugeführt werden.
(1) Studien-spezifischer ausführlicher IC
incl. Beschreibung der Studien-spezifischen Bioprobenverarbeitung und -lagerung in der Biobank
(2) Später optional FREEZE-BC
- für breit angelegte Forschungsprojekte, deren „Herzstück“ Bioproben sind und die i.d.R. in thematisch verwandte Folgeprojekte münden
Bei solchen Forschungsprojekten steht der breite und übergreifende Charakter der Forschungsfrage im Vordergrund (Beispiel: die Untersuchung von entzündlichen Prozessen). Bioproben werden zwar im Rahmen des Forschungsprojektes gewonnen, in der Biobank verarbeitet und asserviert und auch gemäß den FREEZE-Governance-Vorgaben nur nach vorheriger Zustimmung der Studienleitung herausgegeben, jedoch wäre die Probeneinlagerung ohne die Bereitschaft der Spender*innen, übergreifende zukünftige Forschung zu unterstützen, kaum sinnvoll.
(Wichtig: es darf sich nicht um AMG oder Diagnostik-Studien handeln, da Patient*innen die Teilnahme an solchen Studien und damit die möglichen Vorteile bei Diagnostik und Therapie nicht aufgrund des Nichtwillens, den Broad Consent der Biobank zu unterschreiben, verweigert werden darf.)
(1) FREEZE-BC (bindend)
(2) Studien-spezifischer IC
mit Verweis auf FREEZE-BC und Erklärung, warum BC von Bedeutung
- Beratung bei Anträgen an die Ethik-Kommission, insbesondere in Bezug auf Patient*innen-/Proband*inneninformation und –einwilligungserklärung
- Beratung zum Datenschutz
- Beratung zur Beantragung von Biobanking-Leistungen im Kontext von Forschungsanträgen an Drittmittelgeber
- Beratung und Vorbereitungen zur Beantragung und Einrichtung eines FREEZE-Hubs
Mitglied in der GBA

Im Netzwerk die Forschung stärken
Der German Biobank Node (GBN) ist der Dachverband der universitären Biobanken in Deutschland. Unter der Leitung von GBN haben sich 2017 elf Biobankstandorte und zwei IT-Entwicklungszentren in der German Biobank Alliance (GBA) zusammengeschlossen. Sieben weitere Biobankstandorte folgten 2019 und zwei im Jahr 2020. Gemeinsames Ziel ist es, Bioproben und zugehörige Daten für die (inter)nationale biomedizinische Forschung verfügbar zu machen. Dafür harmonisieren die Partner-Biobanken ihr Qualitätsmanagement und vernetzen sich über eine IT-Infrastruktur. Die FREEZE-Biobank ist Partner in diesem Bündnis.
GBN vertritt die Interessen der deutschen Biobanken innerhalb der europäischen Forschungsinfrastruktur BBMRI-ERIC, die sich auf die Vernetzung und den Erfahrungsaustausch von Biobanken innerhalb Europas konzentriert.
Projektauswahl
Publikationen
mit FREEZE-BeteiligungPublikationsauswahl 2024
Analysis of acute COVID-19 including chronic morbidity: protocol for the deep phenotyping National Pandemic Cohort Network in Germany (NAPKON-HAP)
Steinbeis F, Thibeault C, Steinbrecher S, Ahlgrimm Y, Haack IA, August D, Balzuweit B, Bellinghausen C, Berger S, Chaplinskaya-Sobol I, Cornely O, Doeblin P, Endres M, Fink C, Finke C, Frank S, Hanß S, Hartung T, Hellmuth JC, Herold S, Heuschmann P, Heyckendorf J, Heyder R, Hippenstiel S, Hoffmann W, Kelle SU, Knape P, Koehler P, Kretzler L, Leistner DM, Lienau J, Lorbeer R, Lorenz-Depiereux B, Lüttke CD, Mai K, Merle U, Meyer-Arndt LA, Miljukov O, Muenchhoff M, Müller-Plathe M, Neuhann J, Neuhauser H, Nieters A, Otte C, Pape D, Pinto RM, Pley C, Pudszuhn A, Reuken P, Rieg S, Ritter P, Rohde G, Rönnefarth M, Ruzicka M, Schaller J, Schmidt A, Schmidt S, Schwachmeyer V, Schwanitz G, Seeger W, Stahl D, Stobäus N, Stubbe HC, Suttorp N, Temmesfeld B, Thun S, Triller P, Trinkmann F, Vadasz I, Valentin H, Vehreschild M, von Kalle C, von Lilienfeld-Toal M, Weber J, Welte T, Wildberg C, Wizimirski R, Zvork S, Sander LE, Vehreschild J, Zoller T, Kurth F, Witzenrath M. Analysis of acute COVID-19 including chronic morbidity: protocol for the deep phenotyping National Pandemic Cohort Network in Germany (NAPKON-HAP). Infection. 2024 Feb;52(1):93-104. doi: 10.1007/s15010-023-02057-0. Epub 2023 Jul 11. PMID: 37434025; PMCID: PMC10811153.
Single-cell transcriptomics reveal different maturation stages and sublineage commitment of human thymic invariant natural killer T cells
Maas-Bauer K, Köhler N, Stell AV, Zwick M, Acharya S, Rensing-Ehl A, König C, Kroll J, Baker J, Koßmann S, Pradier A, Wang S, Docquier M, Lewis DB, Negrin RS, Simonetta F. Single-cell transcriptomics reveal different maturation stages and sublineage commitment of human thymic invariant natural killer T cells. J Leukoc Biol. 2024 Jan 19;115(2):401-409. doi: 10.1093/jleuko/qiad113. PMID: 37742056; PMCID: PMC10799303.
Non-apoptotic FAS signaling controls mTOR activation and extrafollicular maturation in human B cells
Staniek J, Kalina T, Andrieux G, Boerries M, Janowska I, Fuentes M, Díez P, Bakardjieva M, Stancikova J, Raabe J, Neumann J, Schwenk S, Arpesella L, Stuchly J, Benes V, García Valiente R, Fernández García J, Carsetti R, Piano Mortari E, Catala A, de la Calle O, Sogkas G, Neven B, Rieux-Laucat F, Magerus A, Neth O, Olbrich P, Voll RE, Alsina L, Allende LM, Gonzalez-Granado LI, Böhler C, Thiel J, Venhoff N, Lorenzetti R, Warnatz K, Unger S, Seidl M, Mielenz D, Schneider P, Ehl S, Rensing-Ehl A, Smulski CR, Rizzi M. Non-apoptotic FAS signaling controls mTOR activation and extrafollicular maturation in human B cells. Sci Immunol. 2024 Jan 12;9(91):eadj5948. doi: 10.1126/sciimmunol.adj5948. Epub 2024 Jan 12. PMID: 38215192.
Attenuated effector T cells are linked to control of chronic HBV infection
Heim K, Sagar, Sogukpinar Ö, Llewellyn-Lacey S, Price DA, Emmerich F, Kraft ARM, Cornberg M, Kielbassa S, Knolle P, Wohlleber D, Bengsch B, Boettler T, Neumann-Haefelin C, Thimme R, Hofmann M. Attenuated effector T cells are linked to control of chronic HBV infection. Nat Immunol. 2024 Sep;25(9):1650-1662. doi: 10.1038/s41590-024-01928-4. Epub 2024 Aug 28. PMID: 39198634; PMCID: PMC11362014.
Impaired SARS-CoV-2-Specific CD8+ T Cells After Infection or Vaccination but Robust Hybrid T Cell Immunity in Patients with Multiple Myeloma
Shoumariyeh K, Csernalabics B, Salimi Alizei E, Reinscheid M, Giese S, Ciminski K, Kochs G, Schwemmle M, Lang-Meli J, Maas M, Roehlen N, Karl V, Graeser A, Sogukpinar O, von Metzler I, Grathwohl D, Rasche L, Hebart H, Kull M, Emmerich F, Waller CF, Duyster J, Engelhardt M, Hartmann TN, Bengsch B, Boettler T, Neumann-Haefelin C, Hofmann M, Thimme R, Luxenburger H. Impaired SARS-CoV-2-Specific CD8+ T Cells After Infection or Vaccination but Robust Hybrid T Cell Immunity in Patients with Multiple Myeloma. Vaccines (Basel). 2024 Nov 1;12(11):1249. doi: 10.3390/vaccines12111249. PMID: 39591152; PMCID: PMC11598869.
Publikationsauswahl 2023
Potent high-avidity neutralizing antibodies and T cell responses after COVID-19 vaccination in individuals with B cell lymphoma and multiple myeloma
Keppler-Hafkemeyer A, Greil C, Wratil PR, Shoumariyeh K, Stern M, Hafkemeyer A, Ashok D, Hollaus A, Lupoli G, Priller A, Bischof ML, Ihorst G, Engelhardt M, Marks R, Finke J, Bertrand H, Dächert C, Muenchhoff M, Badell I, Emmerich F, Halder H, Spaeth PM, Knolle PA, Protzer U, von Bergwelt-Baildon M, Duyster J, Hartmann TN, Moosmann A, Keppler OT. Potent high-avidity neutralizing antibodies and T cell responses after COVID-19 vaccination in individuals with B cell lymphoma and multiple myeloma. Nat Cancer. 2023 Jan;4(1):81-95. doi: 10.1038/s43018-022-00502-x. PMID: 36543907; PMCID: PMC9886553.
MAP kinase activating death domain deficiency is a novel cause of impaired lymphocyte cytotoxicity
Schütze K, Groß M, Cornils K, Wustrau K, Schneppenheim S, Lenhartz H, Korenke GC, Janka G, Ledig S, Müller I, Ehl S, Lehmberg K. MAP kinase activating death domain deficiency is a novel cause of impaired lymphocyte cytotoxicity. Blood Adv. 2023 Apr 25;7(8):1531-1535. doi: 10.1182/bloodadvances.2022008195. PMID: 36206192; PMCID: PMC10130601.
Ethical and coordinative challenges in setting up a national cohort study during the COVID-19 pandemic in Germany
Tilch K, Hopff SM, Appel K, Kraus M, Lorenz-Depiereux B, Pilgram L, Anton G, Berger S, Geisler R, Haas K, Illig T, Krefting D, Lorbeer R, Mitrov L, Muenchhoff M, Nauck M, Pley C, Reese JP, Rieg S, Scherer M, Stecher M, Stellbrink C, Valentin H, Winter C, Witzenrath M, Vehreschild JJ. Ethical and coordinative challenges in setting up a national cohort study during the COVID-19 pandemic in Germany. BMC Med Ethics. 2023 Oct 17;24(1):84. doi: 10.1186/s12910-023-00959-0. PMID: 37848886; PMCID: PMC10583323.
Hepatitis D infection induces IFN-β-mediated NK cell activation and TRAIL-dependent cytotoxicity
Groth C, Maric J, Garcés Lázaro I, Hofman T, Zhang Z, Ni Y, Keller F, Seufert I, Hofmann M, Neumann-Haefelin C, Sticht C, Rippe K, Urban S, Cerwenka A. Hepatitis D infection induces IFN-β-mediated NK cell activation and TRAIL-dependent cytotoxicity. Front Immunol. 2023 Dec 8;14:1287367. doi: 10.3389/fimmu.2023.1287367. PMID: 38143742; PMCID: PMC10739304.
Functional Relevance of CTLA4 Variants: an Upgraded Approach to Assess CTLA4-Dependent Transendocytosis by Flow Cytometry
Rojas-Restrepo J, Sindram E, Zenke S, Haberstroh H, Mitsuiki N, Gabrysch A, Huebscher K, Posadas-Cantera S, Krausz M, Kobbe R, Rohr JC, Grimbacher B, Gámez-Díaz L. Functional Relevance of CTLA4 Variants: an Upgraded Approach to Assess CTLA4-Dependent Transendocytosis by Flow Cytometry. J Clin Immunol. 2023 Nov;43(8):2076-2089. doi: 10.1007/s10875-023-01582-9. Epub 2023 Sep 23. Erratum in: J Clin Immunol. 2023 Oct 9;: PMID: 37740092; PMCID: PMC10661720.
Publikationsauswahl 2022
Circulating Tumor DNA Profiling for Detection, Risk Stratification, and Classification of Brain Lymphomas
Mutter JA, Alig SK, Esfahani MS, Lauer EM, Mitschke J, Kurtz DM, Kühn J, Bleul S, Olsen M, Liu CL, Jin MC, Macaulay CW, Neidert N, Volk T, Eisenblaetter M, Rauer S, Heiland DH, Finke J, Duyster J, Wehrle J, Prinz M, Illerhaus G, Reinacher PC, Schorb E, Diehn M, Alizadeh AA, Scherer F. Circulating Tumor DNA Profiling for Detection, Risk Stratification, and Classification of Brain Lymphomas. J Clin Oncol. 2022 Dec 21:JCO2200826. doi: 10.1200/JCO.22.00826. Epub ahead of print. PMID: 36542815.
Long COVID symptoms in exposed and infected children, adolescents and their parents one year after SARS-CoV-2 infection: A prospective observational cohort study
Haddad A, Janda A, Renk H, Stich M, Frieh P, Kaier K, Lohrmann F, Nieters A, Willems A, Huzly D, Dulovic A, Schneiderhan-Marra N, Jacobsen EM, Fabricius D, Zernickel M, Stamminger T, Bode SFN, Himpel T, Remppis J, Engel C, Peter A, Ganzenmueller T, Hoffmann GF, Haase B, Kräusslich HG, Müller B, Franz AR, Debatin KM, Tönshoff B, Henneke P, Elling R. Long COVID symptoms in exposed and infected children, adolescents and their parents one year after SARS-CoV-2 infection: A prospective observational cohort study. EBioMedicine. 2022 Oct;84:104245. doi: 10.1016/j.ebiom.2022.104245. Epub 2022 Sep 22. PMID: 36155957; PMCID: PMC9495281.
SARS-CoV-2-specific T-cell epitope repertoire in convalescent and mRNA-vaccinated individuals
Lang-Meli J, Luxenburger H, Wild K, Karl V, Oberhardt V, Salimi Alizei E, Graeser A, Reinscheid M, Roehlen N, Reeg DB, Giese S, Ciminski K, Götz V, August D, Rieg S, Waller CF, Wengenmayer T, Staudacher D, Huzly D, Bengsch B, Kochs G, Schwemmle M, Emmerich F, Boettler T, Thimme R, Hofmann M, Neumann-Haefelin C. SARS-CoV-2-specific T-cell epitope repertoire in convalescent and mRNA-vaccinated individuals. Nat Microbiol. 2022 May;7(5):675-679. doi: 10.1038/s41564-022-01106-y. Epub 2022 Apr 28. PMID: 35484232; PMCID: PMC9064790.
COVID-19 mRNA booster vaccine induces transient CD8+ T effector cell responses while conserving the memory pool for subsequent reactivation
Reinscheid M, Luxenburger H, Karl V, Graeser A, Giese S, Ciminski K, Reeg DB, Oberhardt V, Roehlen N, Lang-Meli J, Heim K, Gross N, Baum C, Rieg S, Speer C, Emmerich F, Breisinger S, Steinmann D, Bengsch B, Boettler T, Kochs G, Schwemmle M, Thimme R, Neumann-Haefelin C, Hofmann M. COVID-19 mRNA booster vaccine induces transient CD8+ T effector cell responses while conserving the memory pool for subsequent reactivation. Nat Commun. 2022 Aug 8;13(1):4631. doi: 10.1038/s41467-022-32324-x. PMID: 35941157; PMCID: PMC9358914.
Publikationsauswahl 2021
The expansion of human T-bet high CD21 low B cells is T cell dependent
Keller B, Strohmeier V, Harder I, Unger S, Payne KJ, Andrieux G, Boerries M, Felixberger PT, Landry JJM, Nieters A, Rensing-Ehl A, Salzer U, Frede N, Usadel S, Elling R, Speckmann C, Hainmann I, Ralph E, Gilmour K, Wentink MWJ, van der Burg M, Kuehn HS, Rosenzweig SD, Kölsch U, von Bernuth H, Kaiser-Labusch P, Gothe F, Hambleton S, Vlagea AD, Garcia Garcia A, Alsina L, Markelj G, Avcin T, Vasconcelos J, Guedes M, Ding JY, Ku CL, Shadur B, Avery DT, Venhoff N, Thiel J, Becker H, Erazo-Borrás L, Trujillo-Vargas CM, Franco JL, Fieschi C, Okada S, Gray PE, Uzel G, Casanova JL, Fliegauf M, Grimbacher B, Eibel H, Ehl S, Voll RE, Rizzi M, Stepensky P, Benes V, Ma CS, Bossen C, Tangye SG, Warnatz K. The expansion of human T-bethighCD21low B cells is T cell dependent. Sci Immunol. 2021 Oct 15;6(64):eabh0891. doi: 10.1126/sciimmunol.abh0891. Epub 2021 Oct 8. PMID: 34623902.
A distinct CD38+CD45RA+ population of CD4+, CD8+, and double-negative T cells is controlled by FAS
Maccari ME, Fuchs S, Kury P, Andrieux G, Völkl S, Bengsch B, Lorenz MR, Heeg M, Rohr J, Jägle S, Castro CN, Groß M, Warthorst U, König C, Fuchs I, Speckmann C, Thalhammer J, Kapp FG, Seidel MG, Dückers G, Schönberger S, Schütz C, Führer M, Kobbe R, Holzinger D, Klemann C, Smisek P, Owens S, Horneff G, Kolb R, Naumann-Bartsch N, Miano M, Staniek J, Rizzi M, Kalina T, Schneider P, Erxleben A, Backofen R, Ekici A, Niemeyer CM, Warnatz K, Grimbacher B, Eibel H, Mackensen A, Frei AP, Schwarz K, Boerries M, Ehl S, Rensing-Ehl A. A distinct CD38+CD45RA+ population of CD4+, CD8+, and double-negative T cells is controlled by FAS. J Exp Med. 2021 Feb 1;218(2):e20192191. doi: 10.1084/jem.20192191. PMID: 33170215; PMCID: PMC7658692.
Characterization of pre-existing and induced SARS-CoV-2-specific CD8 + T cells
Schulien I, Kemming J, Oberhardt V, Wild K, Seidel LM, Killmer S, Sagar, Daul F, Salvat Lago M, Decker A, Luxenburger H, Binder B, Bettinger D, Sogukpinar O, Rieg S, Panning M, Huzly D, Schwemmle M, Kochs G, Waller CF, Nieters A, Duerschmied D, Emmerich F, Mei HE, Schulz AR, Llewellyn-Lacey S, Price DA, Boettler T, Bengsch B, Thimme R, Hofmann M, Neumann-Haefelin C. Characterization of pre-existing and induced SARS-CoV-2-specific CD8+ T cells. Nat Med. 2021 Jan;27(1):78-85. doi: 10.1038/s41591-020-01143-2. Epub 2020 Nov 12. PMID: 33184509.
Anti-platelet factor 4 antibodies causing VITT do not cross-react with SARS-CoV-2 spike protein
Greinacher A, Selleng K, Mayerle J, Palankar R, Wesche J, Reiche S, Aebischer A, Warkentin TE, Muenchhoff M, Hellmuth JC, Keppler OT, Duerschmied D, Lother A, Rieg S, Gawaz MP, Mueller KAL, Scheer CS, Napp M, Hahnenkamp K, Lucchese G, Vogelgesang A, Flöel A, Lovreglio P, Stufano A, Marschalek R, Thiele T; Immune-Response in COVID-19 Vaccination Study Group. Anti-platelet factor 4 antibodies causing VITT do not cross-react with SARS-CoV-2 spike protein. Blood. 2021 Oct 7;138(14):1269-1277. doi: 10.1182/blood.2021012938. PMID: 34280256; PMCID: PMC8294553.
Robust and durable serological response following pediatric SARS-CoV-2 infection
Renk H, Dulovic A, Seidel A, Becker M, Fabricius D, Zernickel M, Junker D, Groß R, Müller J, Hilger A, Bode SFN, Fritsch L, Frieh P, Haddad A, Görne T, Remppis J, Ganzemueller T, Dietz A, Huzly D, Hengel H, Kaier K, Weber S, Jacobsen EM, Kaiser PD, Traenkle B, Rothbauer U, Stich M, Tönshoff B, Hoffmann GF, Müller B, Ludwig C, Jahrsdörfer B, Schrezenmeier H, Peter A, Hörber S, Iftner T, Münch J, Stamminger T, Groß HJ, Wolkewitz M, Engel C, Liu W, Rizzi M, Hahn BH, Henneke P, Franz AR, Debatin KM, Schneiderhan-Marra N, Janda A, Elling R. Robust and durable serological response following pediatric SARS-CoV-2 infection. Nat Commun. 2022 Jan 10;13(1):128. doi: 10.1038/s41467-021-27595-9. PMID: 35013206; PMCID: PMC8748910.